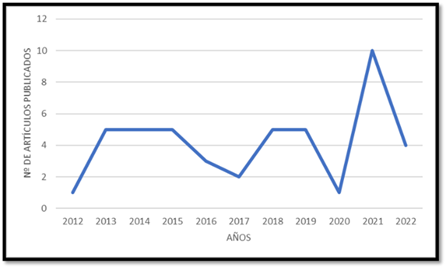
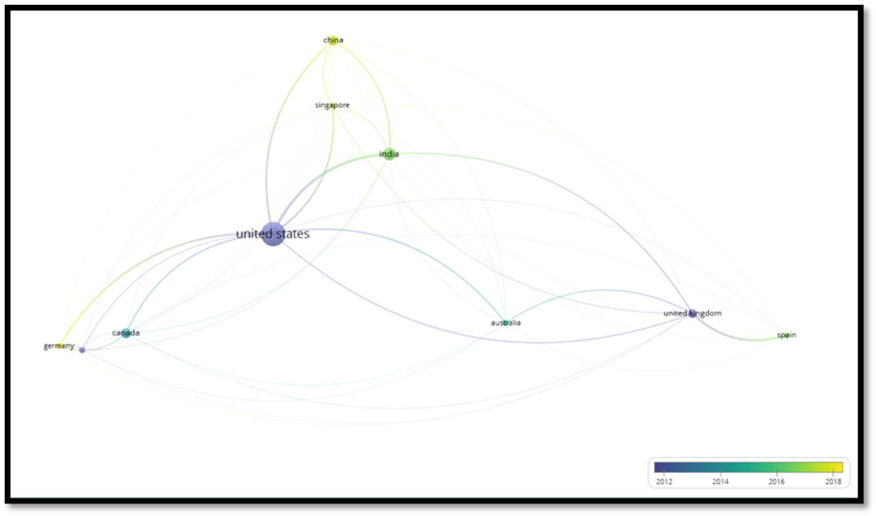
CONCLUSIONES
Las investigaciones en métodos de
detección de coliformes totales en el agua, es de utilidad para la población,
esto ayudará a elaborar una técnica útil para evaluaciones microbiológicas en
corto tiempo. En el Perú se evidencia un vacío de conocimiento acerca de
métodos recomendables para utilizar en el análisis de calidad microbiológica de
agua, a comparación de Estados Unidos, que es un país líder en métodos
moleculares.
REFERENCIAS BIBLIOGRÁFICAS
Amidoun, S. (2017). Escherichia coli Recent Advances on Physiology,
Pathogenesis and Biotechnological Applications.
Fusco,
A., Batista, K., Oliveira, C., & Brito, E. (2010). Desenvolvimento de PCR
multiplex para detecção e diferenciação de categorias de Escherichia coli
diarreiogênicas. Revista Pan-Amazônica de Saúde, 1(2). https://doi.org/10.5123/s2176-6223201000020000
Girones,
R., Ferrús, M. A., Alonso, J. L., Rodriguez-Manzano, J., Calgua, B., de Abreu
Corrêa, A., Hundesa, A., Carratala, A., & Bofill-Mas, S. (2010). Molecular detection of
pathogens in water - The pros and cons of molecular techniques. In Water
Research (Vol. 44, Issue 15, pp. 4325–4339). Elsevier Ltd.
https://doi.org/10.1016/j.watres.2010.06.030
Mendes, D., & Domingues, L. (2015). On the track for an efficient
detection of Escherichia coli in water: A review on PCR-based methods. In
Ecotoxicology and Environmental Safety (Vol. 113, pp. 400–411). Academic Press.
https://doi.org/10.1016/j.ecoenv.2014.12.015
Moyano,
S., & Marín, G. (2014). Técnica de filtración ISO 9308 aplicada al
monitoreo de agua de red. RADI, 450(5900), 53–58.
Md Khudzari, J., Kurian, J., Tartakovsky, B., & Raghavan, G. S. V.
(2018). Bibliometric analysis of global research trends on microbial fuel cells
using Scopus database. Biochemical Engineering Journal, 136, 51–60.
https://doi.org/10.1016/j.bej.2018.05.002
Nurliyana, M. R., Sahdan, M. Z., Wibowo, K. M., Muslihati, A., Saim, H.,
Ahmad, S. A., Sari, Y., & Mansor, Z. (2018). The Detection Method of
Escherichia coli in Water Resources: A Review. Journal of Physics: Conference
Series, 995(1). https://doi.org/10.1088/1742-6596/995/1/012065
Salaz R (2019). Métodos
de búsqueda de información Bibliográfica
Silva,
R., Rocha, R. S., Ramos, G. L. P. A., Xavier-Santos, D., Pimentel, T. C.,
Lorenzo, J. M., Henrique Campelo, P., Cristina Silva, M., Esmerino, E. A.,
Freitas, M. Q., & Cruz, A. G. (2022). What are the challenges for ohmic
heating in the food industry? Insights of a bibliometric analysis. Food
Research International, 157(March).
https://doi.org/10.1016/j.foodres.2022.111272
Maguire
M., Kase J.A., Brown E.W., Allard M.W., Musser S.M., González-Escalona N
(2022). Metagenomic
survey of agricultural water using long read sequencing: Considerations for a
successful análisis.
Treebupachatsakul T., Lochotinunt C., Teechot T., Pensupa N., Pechprasarn
S.(2022). Gelatin-Based Microfluidic Channel for Quantitative E. Coli Detection
Using Blue Fluorescence of 4-Methyl-Umbelliferone Product and a Smartphone
Camera
Rishi M., Amreen K., Mohan J.M., Javed A., Dubey S.K., Goel S. (2002).
Rapid, sensitive and specific electrochemical detection of E. coli using
graphitized mesoporous carbon-modified electrodes
Zarrinkhat F., Jofre-Roca L., Jofre M., Rius J.M., Romeu J. (2022).
Experimental Verification of Dielectric Models with a Capacitive Wheatstone
Bridge Biosensor for Living Cells: E. coli
Saez
J., Catalan-Carrio R., Owens R.M., Basabe-Desmonts L., Benito-Lopez F. (2021). Microfluidics and
materials for smart water monitoring: A review
Rani A., Ravindran V.B., Surapaneni A., Mantri N., Ball A.S. (2021)
Review: Trends in point-of-care diagnosis for Escherichia coli O157:H7 in food
and wáter.
Khan I.U.H., Becker A., Cloutier M., Plötz M., Lapen D.R., Wilkes G.,
Topp E., Abdulmawjood A. (2021). Loop-mediated isothermal amplification:
Development, validation and application of simple and rapid assays for
quantitative detection of species of Arcobacteraceae family- and
species-specific Aliarcobacter faecis and Aliarcobacter lanthieri.
Shaik S., Saminathan A., Sharma D., Krishnaswamy J.A., Mahapatra
D.R.(2021) Monitoring microbial growth on a microfluidic lab-on-chip with
electrochemical impedance spectroscopic technique.
Vishwakarma
A., Lal R., Ramya M. (2021). Aptamer-based approaches for the detection of waterborne
pathogens.
Olalemi A.O., Ige O.M., James G.A., Obasoro F.I., Okoko F.O., Ogunleye
C.O. (2021). Detection of enteric bacteria in t w o groundwater sources a n d
associated microbial health risks.
Demoliner M., Gularte J.S., Girardi V., Eisen A.K.A., de Souza F.G.,
Staggemeier R., Henzel A., Spilki F.R. (2021). Microbial Source Tracking in
Small Farms: Use of Different Methods for Adenovirus Detection
Gangar
T., Satyam K., Patra S. (2021). Monitoring/sensing techniques to address pollutant
heterogeneity assessment in wastewater.
Kora A.J. (2021). Zirconium alginate beads: A renewable source for the
biosorption of fluoride from contaminated ground wáter.
Ward J.S.T., Lapworth D.J., Read D.S., Pedley S., Banda S.T., Monjerezi
M., Gwengweya G., MacDonald A.M. (2021). Tryptophan-like fluorescence as a
high-level screening tool for detecting microbial contamination in drinking
wáter.
Khan F.M., Gupta R. (2020). Escherichia coli (e. coli) as an indicator of
fecal contamination in groundwater: A review.
Bigham T., Dooley J.S.G., Ternan N.G., Snelling W.J., Héctor Castelán
M.C., Davis J. (2019). Assessing microbial water quality: Electroanalytical
approaches to the detection of coliforms.
Wolf-Baca M., Siedlecka A. (2019). Detection of pathogenic bacteria in
hot tap water using the qPCR method: preliminary research.
Han
E.J.Y., Palanisamy K., Hinks J., Wuertz S. (2019). Parameter selection for a microvolume
electrochemical Escherichia coli detector for pairing with a concentration
device.
Loo A., Bivins A., John V., Becker S., Evanchec S., George A., Hernandez
V., Mullaney J., Tolentino L., Yoo R., Nagarnaik P., Labhasetwar P., Brown
J.(2019). Development and field testing of low-cost, quantal microbial assays
with volunteer reporting as scalable means of drinking water safety estimation.
Lacey R.F., Ye D., Ruffing A.M. (2019). Engineering and characterization
of copper and gold sensors in Escherichia coli and Synechococcus sp. PCC 7002.
Malec A., Kokkinis G., Haiden C., Giouroudi I. (2018). Biosensing system
for concentration quantification of magnetically labeled e. Coli in water
simples.
Wu G., Meyyappan M., Lai K.W.C. (2018). Simulation of graphene
field-effect transistor biosensors for bacterial detection,
Yin H.-B., Patel J.(2018). Comparison of methods to determine the
microbial quality of alternative irrigation Waters,
Ozeh U.O., Nnanna A.G.A., Ndukaife J.C. (2018). Coupling
immunofluorescence and optoelectrokinetic technique for Escherichia coli
detection and quantification in wáter.
Gutiérrez-del-Río
I., Marín L., Fernández J., Millán M.Á.S., Ferrero F.J., Valledor M., Campo
J.C., Cobián N., Méndez I., Lombó F. (2018). Development of a biosensor protein
bullet as a fluorescent method for fast detection of Escherichia coli in
drinking wáter
Gunda
N.S.K., Dasgupta S., Mitra S.K. (2017). DipTest: A litmus test for E. coli
detection in wáter.
Kheiri R., Ranjbar R., Memariani M., Akhtari L. (2017). Multiplex PCR for
detection of water-borne bacteria.
Eltzov E., Marks R.S. (2016). Miniaturized Flow Stacked Immunoassay for
Detecting Escherichia coli in a Single Step.
Yang
X., Yang K., Luo Y., Fu W. (2016). Terahertz spectroscopy for bacterial detection:
opportunities and challenges.
Shaibani P.M., Jiang K., Haghighat G., Hassanpourfard M., Etayash H.,
Naicker S., Thundat T. (2016). The detection of Escherichia coli (E. coli) with
the pH sensitive hydrogel nanofiber-light addressable potentiometric sensor
(NF-LAPS).
Ramasamy M., Yi D.K., An S.S.A. (2015). Enhanced detection sensitivity of
escherichia coli 0157:H7 using surface–modified gold nanorods.
Bridgeman J., Baker A., Brown D., Boxall J.B. (2015). Portable LED
fluorescence instrumentation for the rapid assessment of potable water quality.
Nigam V.K., Shukla P. (2015). Enzyme based biosensors for detection of
environmental pollutants-A review.
Saxena T., Kaushik P., Krishna Mohan M. (2015). Prevalence of E. coli
O157: H7 in water sources: An overview on associated diseases, outbreaks and
detection methods.
Nicolini A.M., Fronczek C.F., Yoon J.-Y. (2015). Droplet-based
immunoassay on a 'sticky' nanofibrous surface for multiplexed and dual
detection of bacteria using smartphones.
Gomi
R., Matsuda T., Matsui Y., Yoneda M. (2014). Fecal source tracking in water by
next-generation sequencing technologies using host-specific escherichia coli
genetic markers.
Bari M.L., Yeasmin S. (2014). Water Quality Assessment: Modern
Microbiological Techniques.
Loff M., Mare L., De Kwaadsteniet M., Khan W. (2014). 3M™ Molecular
Detection system versus MALDI-TOF mass spectrometry and molecular techniques
for the identification of Escherichia coli 0157: H7, Salmonella spp. &
Listeria spp
Stauber C., Miller C., Cantrell B., Kroell K. (2014). Evaluation of the
compartment bag test for the detection of Escherichia coli in wáter.
Gonzalez R.A.,
Noble R.T. (2014). Comparisons of statistical models to predict fecal indicator bacteria
concentrations enumerated byqPCR- and culture-based methods.
Kim T., Han J.-I. (2013). Fast detection and quantification of
Escherichia coli using the base principle of the microbial fuel cell.
Durso L.M. (2013). Primary isolation of shiga toxigenic escherichia coli
from environmental sources.
Sbodio
A., Maeda S., Lopez-Velasco G., Suslow T.V. (2013). Modified Moore swab optimization and
validation in capturing E. Coli O157: H7 and Salmonella enterica in large
volume field samples of irrigation wáter.
Nagalambika C., Murthy S.M. (2013). Revalidation of testing methods for
assessing microbial safety of groundwater.
Wandermur G.L., Rodrigues D.M.C., Queiroz V.M., Gonçalves M.N., Miguel
M.A.L., Werneck M.M., Allil R.C.S.B. (2013). Development of an immunosensor of
plastic optical fiber for detection of microorganisms in water and
environmental monitoring.
McMahan L., Grunden A.M., Devine A.A., Sobsey M.D. (2012). Evaluation of
a quantitative H2S MPN test for fecal microbes analysis of water using
biochemical and molecular identification.
![]() *
*